Dell EMC Ready Solution for HPC Life Sciences: Pipeline da Tuxedo com CPU Cascade Lake e atualização do Lustre/ME4
概要: Este artigo aborda o white paper intitulado "Dell EMC Ready Solution for HPC Life Sciences: Pipeline da Tuxedo com CPU Cascade Lake e atualização do Lustre/ME4".
手順
Nota: Artigo escrito por Kihoon Yoon do HPC e AI Innovation Lab em dezembro de 2019
O novo hardware e o pipeline atualizado juntos aumentam o throughput três vezes mais em relação à Ready Solution anterior.
Visão geral
A análise de expressão gênica é tão importante quanto identificar o Polimorfismo de nucleotídeo único (SNP), inserção/exclusão (indel) ou reestruturação cromossômica. Eventualmente, todos os eventos fisiológicos e bioquímicos dependem dos produtos finais da expressão gênica, as proteínas. Embora a maioria dos mamíferos tenha uma camada de controle adicional antes da expressão de proteínas, saber quantas transcrições existem em um sistema ajuda a caracterizar o status bioquímico de uma célula. Idealmente, uma tecnologia que nos permita quantificar todas as proteínas em uma célula poderia se destacar no progresso das Ciências da Vida de maneira significativa, no entanto, estamos longe de conseguir isso.
Aqui, neste blog, testamos um popular pipeline de análise de dados RNA-Seq conhecido como pipeline da Tuxedo (1). A suíte de pipelines Tuxedo
 Figura 1: atualização do Pipeline da Tuxedo com a etapa Cuffquant
Figura 1: atualização do Pipeline da Tuxedo com a etapa Cuffquant
As configurações do cluster de teste estão resumidas na Tabela 1.
| Tabela 1: configuração testada do nó de computação |
|
| Dell EMC PowerEdge C6420 |
|
| CPU |
2x Xeon® Gold 6248 20c 2,5 GHz (Cascade Lake) |
| RAM |
12x 16 GB a 2933 MT/s |
| OS |
RHEL 7.6 |
| Interconexão |
Intel® Omni-Path |
| Perfil do sistema BIOS |
Desempenho otimizado |
| Processador lógico |
Disabled |
| Tecnologia de virtualização |
Disabled |
| tophat |
2.1.1 |
| bowtie2 |
2.2.5 |
| R |
3.6 |
| bioconductor-cummerbund |
2.26.0 |
Os nós de computação testados foram conectados à Dell EMC Ready Solution for Lustre Storage por meio do Intel® Omni-Path (3). A configuração resumida do armazenamento está listada na Tabela 2.
| Tabela 2: especificações de hardware e software da solução de armazenamento Lustre |
|
| Dell EMC Ready Solution for Lustre Storage |
|
| Número de nós |
1x Dell EMC PowerEdge R640 como Integrated Manager for Lustre (IML) |
| Processadores |
Servidor IML: Intel Xeon Gold 5118 duplo a 2,3 GHz |
| Memória |
Servidor IML: 12x RDIMMs DDR4 de 8 GB e 2.666 MT/s |
| Controladores de armazenamento |
2x HBAs SAS Dell de 12 Gb/s (em cada MDS) |
| Compartimentos |
4x ME4084 com um total de 336 discos rígidos SAS NL de 7.200 RPM de 8 TB |
| Compartimento |
1x ME4024 com 24x SSDs SAS de 960 GB. Compatível com até 4,688 bilhões de arquivos/inodes |
| Controladores RAID |
RAID duplex nos compartimentos ME4084 e ME4024 |
| Sistema operacional |
CentOS 7.5 x86_64 |
| Versão do kernel |
3.10.0-862.el7.x86_64 |
| Versão do BIOS |
1.4.5 |
| Intel Omni-Path |
10.8.0.0 |
| File system Lustre |
2.10.4 |
| Versão de IML |
4.0.7.0 |
Um estudo de desempenho do pipeline RNA-Seq não é trivial porque o fluxo de trabalho natural requer arquivos de entrada não idênticos. 185 conjuntos de dados de leitura em pares do RNA-Seq são coletados de um repositório de dados público
Avaliação de Desempenho
Teste de duas amostras
Na Figura 2, o tempo de execução de cada etapa é representado. O teste foi executado em dois nós de computação com duas amostras contendo aproximadamente 25 milhões de dados de leitura de RNA-Seq. A etapa Tophat é iniciada para cada amostra em um nó de computação em paralelo. Posteriormente, Cufflinks começa após a conclusão de Tophat. A etapa Cuffmerge combina os resultados das duas execuções do Cufflinks. A etapa Cuffquant é adicionada para quantificar a expressão de genes em cada amostra, e os resultados são examinados mais adiante nas etapas Cuffdiff e Cuffnorm. A última etapa, CummeRbund, é uma etapa de análise estatística do pacote R do CummeRbund e gera um relatório visualizado, conforme mostrado na Figura 2. Figura 2 Tempo de execução total do pipeline da Tuxedo com duas amostras: SRR1608490 e SRR934809.
Figura 2 Tempo de execução total do pipeline da Tuxedo com duas amostras: SRR1608490 e SRR934809.
A Figura 3 mostra genes expressos diferencialmente de 8 amostras executadas (cada amostra consiste em 4 cópias) em vermelho com valores de p significativamente menores (eixo Y) em comparação com outras expressões gênicas ilustradas em preto1. O eixo X representa as alterações de dobra na base logarítmica de 2, e essas alterações de dobra de cada gene são plotadas em relação aos valores de p. Mais amostras trarão uma melhor estimativa da expressão gênica. O gráfico superior direito são expressões gênicas na amostra 2 em comparação com a amostra 1, enquanto o gráfico inferior esquerdo são expressões gênicas na amostra 1 em comparação com a amostra 2. A expressão gênica nos pontos pretos não é significativamente diferente em ambas as amostras. 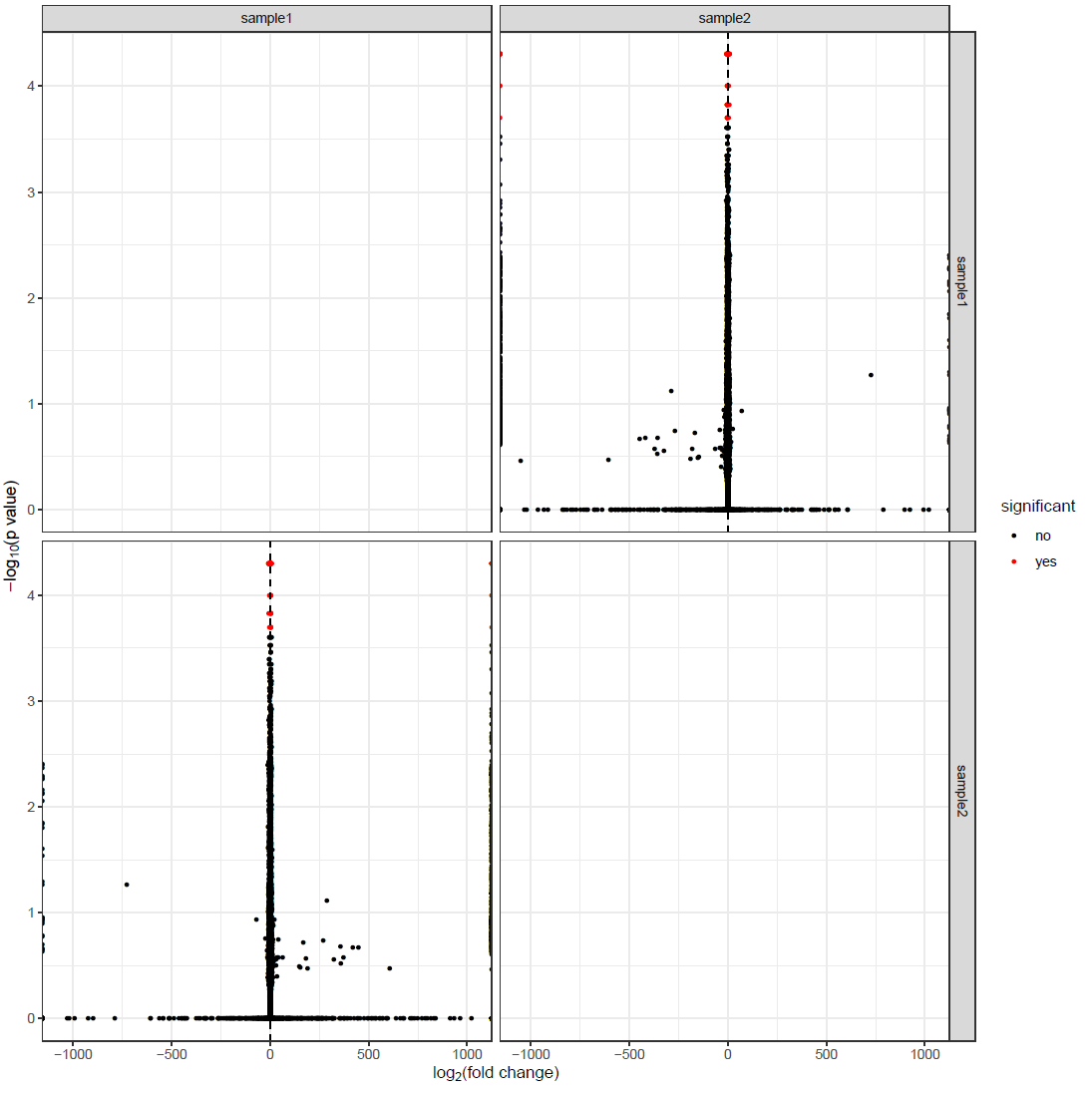 Figura 3: gráfico de vulcão dos resultados de Cuffdiff
Figura 3: gráfico de vulcão dos resultados de Cuffdiff
Teste de throughput: pipeline único com mais de duas amostras, cópias biológicas e técnicas
Os estudos típicos de RNA-Seq consistem em múltiplas amostras, às vezes centenas de amostras diferentes, amostras normais versus amostras com doença ou amostras não tratadas versus tratadas. Essas amostras costumam apresentar alto nível de ruído devido a suas razões biológicas; portanto, a análise requer um procedimento vigoroso de pré-processamento de dados.
Testamos várias quantidades de amostras (todos os dados diferentes de RNA-Seq selecionados do conjunto de dados de 185 leituras em pares) para ver a quantidade de dados que pode ser processada por 8 nós em um cluster do PowerEdge C6420. Como mostrado na Figura 4, os tempos de execução com 2, 4, 8, 16, 32 e 64 amostras crescem exponencialmente quando o número de amostras aumenta. O número de bilhões de fragmentos/dia aumentou quase três vezes com o armazenamento do Cascade Lake 6248/LustreME4 e o pipeline atualizado. Figura 4: comparações de throughput com 8x C6420s entre o Cascade Lake 6248/LustreME4 e Skylake 6148/H600
Figura 4: comparações de throughput com 8x C6420s entre o Cascade Lake 6248/LustreME4 e Skylake 6148/H600
A etapa Cuffmerge não diminui à medida que o número de amostras aumenta, enquanto as etapas Cuffdiff e Cuffnorm diminuem significativamente. Especialmente, a etapa Cuffdiff torna-se um gargalo para o pipeline, uma vez que o tempo de execução cresce exponencialmente (Figura 5). Embora o tempo de execução do Cuffnorm aumente exponencialmente como o Cuffdiff, ele é passível de ser ignorado, pois o tempo de execução do Cuffnorm é limitado pelo tempo de execução do Cuffdiff. A adição da etapa Cuffquant melhorou significativamente o tempo de execução do Cuffdiff. 30 horas de redução do tempo de execução na etapa Cuffdiff, e o Cuffnorm foi concluído 20 horas mais rápido com a etapa Cuffquant. Embora o ganho de desempenho de Cuffnorm não seja visível, uma vez que Cuffdiff e Cuffnorm começam ao mesmo tempo.
Figura 5 Incremento do tempo de execução no Cuffdiff e no Cuffnorm
Conclusão
Os resultados do teste de throughput mostram que o PowerEdge C6420s de 8 nós com o armazenamento Lustre pode processar aproximadamente 2,7 bilhões de fragmentos de 64 amostras com cerca de 50 milhões de leituras pareadas cada (25 MF) por meio do pipeline da Tuxedo ilustrado na Figura 1. Como o Pipeline da Tuxedo é relativamente mais rápido do que outros pipelines populares, é difícil generalizar ou utilizar esses resultados para dimensionar um sistema HPC com precisão. No entanto, os resultados podem ajudar a fazer uma estimativa aproximada do tamanho do sistema de HPC.
Recursos
1. Expressão gênica diferencial RNA-Seq: Tutorial básico. [Online] https://melbournebioinformatics.github.io/MelBioInf_docs/tutorials/rna_seq_dge_basic/rna_seq_basic_tuxedo/.
2. Referência de desempenho de pipeline RNA-Seq com o Dell EMC Ready Bundle for HPC Life Sciences. [On-line] https://downloads.dell.com/manuals/all-products/esuprt_software/esuprt_it_ops_datcentr_mgmt/high-computing-solution-resources_white-papers86_en-us.pdf.
3. Dell EMC Ready Solution for HPC Lustre Storage. [Link inativo em 07/2024]
Nota: estes são selecionados aleatoriamente a partir de um conjunto de amostras sem quaisquer associações significativas entre eles.