Acceleration af genomikdataanalyse med NVIDIA Clara Parabricks med Dell EMC DSS 8440-serveren og NVIDIA T4 GPU'er
Summary: Denne artikel indeholder oplysninger om at accelerere genomikdataanalyse ved brug af NVIDIA Parabricks på Dell EMC DSS 8440 med NVIDIA T4 GPU'er.
This article applies to
This article does not apply to
This article is not tied to any specific product.
Not all product versions are identified in this article.
Instructions
Oversigt
Det første trin til behandling af NGS-data (Next Generation Sequencing) kaldes primær analyse. Dette trin er specifikt for sekventeringsinstrumentet og genererer flere FASTQ-filer, der indeholder sekventeringslæsninger. I det næste trin, kaldet sekundær analyse, tilknyttes FASTQ-sekventeringslæsningerne til et referencegenom eller et referencetranskriptom. Yderligere behandling identificerer varianter eller forskelle mellem den interessante prøve og en reference. Varianterne annoteres og fortolkes i efterfølgende downstream-trin. Den sekundære analyses varighed for en enkelt prøve spænder fra få timer til flere dage, alt afhængigt af datastørrelse, tilgængelige databehandlingsressourcer, softwaren og det analytiske workflow.Sekundær analyse er en databehandlings- og lagringskrævende proces, især når du behandler hundredvis til tusindvis af genomer. Der findes mange strategier for at undgå flaskehalse i sekundære analyser. Indtil for nylig er behovet for at implementere hardwareacceleration ved hjælp af GPU'er eller FPGA'er forblevet lavt takket være tilpasset software, der kræves af hardwareacceleratorer. Parabricks' genomiksoftware, som blev overtaget af NVIDIA i 2019, er den første til at anvende en softwarestak, der udfører forskellige genomikanalysearbejdsprocesser med GPU'er. Vi har testet Parabricks med Dell EMC PowerEdge C4140/4x NVIDIA® Tesla® V100 GPU'er for omkring to år siden. Dell introducerede mange teknologiske fremskridt i deres servere og lagringsløsninger, og NVIDIA Clara Parabricks har udgivet stabile versioner med forbedret acceleration og tilføjelsen af variantbestemmelse. F.eks. så et multi-GPU-serverdesign, der er baseret på Dell EMC DSS 8440-serveren med NVIDIA® Tesla® T4 GPU'er, lovende ud i forhold til at accelerere den sekundære analyse, samtidig med at det tilbød en attraktiv balance mellem pris og ydeevne. Denne blog rapporterer en ny referencearkitektur og benchmark-resultater for sekundær analyse med NVIDIA Clara Parabricks på en multi-Tesla® T4 GPU, DSS 8440-server med Dell EMC Isilon F800-lagring.
Referencearkitektur
Figur 1 illustrerer den testede referencearkitektur. Arkitekturen er modulopbygget og nem at skalere. NVIDIA Clara Parabricks-applikationssoftwaren bruger en eller flere GPU'er, der gør udskalering så enkelt som muligt. Hardwarebyggeklodserne består af Dell EMC PowerEdge R640 som en administrationsnode, DSS 8440-serveren til GPU-databehandling samt Dell EMC Isilon F800-lagring.
Figur 1 Testet referencearkitektur
DSS 8440, 2 sokler, 4U-serveren kan tage op til 10 brancheførende NVIDIA® Tesla® V100S Tensor Core GPU'er, op til 10 NVIDIA® Quadro RTX™ GPU'er eller op til 16 NVIDIA-Tesla T4 GPU'er, der giver en enorm kraft. Den detaljerede konfiguration af DSS 8440 er anført i tabel 1.
| Dell EMC DSS 8440 | |
|---|---|
| CPU | 2x Xeon® Gold 6248R, 24 kerner 3,0 GHz |
| RAM | 24x 64 GB ved 2933 MTps |
| Operativsystem | Red Hat Enterprise Linux Server version 7.4 (Maipo) |
| BIOS-systemprofil | Ydeevneoptimeret |
| Logisk processor | Deaktiveret |
| Virtualiseringsteknologi | Deaktiveret |
| Acceleratorer | 16x NVIDIA® Tesla® T4 GPU'er |
| Parabricks | v3.0.0.05 |
To Z9100-ON-switche leverede sammenkoblingen mellem databehandlingsnoden og Isilon F800-lagringsklyngen. En ekstra switch, N2248X-ON, bruges til administration.
NGS-data
Data til benchmarking af driftstiden for sekundær analyse bestod af tre datasæt for menneskelig helgenom-sekventering (WGS), ERR091571, SRR3124837 og ERR194161, som repræsenterer henholdsvis 10x, 30x og 50x prøvedækning. Disse datasæt er tilgængelige i Det Europæiske Nukleotid-arkiv, ENA (European Nucleotide Archive).Ydeevneevaluering
Softwareforbedringer reducerer driftstidenNVIDIA fortsætter med at introducere softwareforbedringer til NVIDIA Clara Parabricks. Figur 2 viser reduktionen af driftstiden mellem to versioner af Parabricks, der udfører kimcelle-pipelinen vha. testmiljøet med en Dell PowerEdge C4140-server med 4x V100 GPU'er. Flytningen fra v2.1.0 til v3.0.0 reducerede driftstiden med 42 %.

Figur 2 driftstid for seneste version af Parabricks' kimcellevariantbestemmelses-pipeline.
Ydeevne for DSS 8440 med 16x T4s
driftstiden for en sekundær analyse med NVIDIA Clara Parabricks ved brug af en enkelt T4 GPU er ca. 30 % langsommere end ved brug af én V100 GPU. Men to (2) T4 GPU'er giver ca. 10 % mere TFLOPS end én (1) V100 GPU med ca. de halve omkostninger. DSS 8440 leverer op til 16 PCIe-slots, der åbner mulighed for at designe en T4 GPU-baseret server, der leverer en driftstidsydeevne svarende til et C4140-system med fire V100 GPU'er, men med lavere omkostninger.Parabricks-kimcelleanalysen blev udført ved brug af en PowerEdge DSS 8440 med 16 T4 GPU'er. For hvert WGS-prøvedatasæt, som blev beskrevet tidligere, blev driftstiden registreret ved brug af 1, 2, 4, 8 og 16 T4 GPU'er pr. sekundær analyse. Resultaterne er afbildet i Figur 3 til 5. Overordnet skalerer driftstiden ikke lineært, når antallet af GPU'er pr. analyse øges. Skaleringsmønsteret svarer til øgningen af datamængden pr. prøve fra 10x til 50x dækning.
Selvom den ikke præsenteres her, skalerede en tidligere Dell EMC-undersøgelse af Parabricks-driftstidsresultater ved brug af otte eller flere V100 GPU'er pr. analyse ikke så effektivt som T4 GPU'erne. Yderligere test påviste, at 6 T4 GPU'er genererede driftstidsresultater, der var næsten identiske med 4 V100 GPU'er.

Figur 3 Sammenligninger af ydeevne med 10x WGS

Figur 4 Sammenligninger af ydeevne med 30x WGS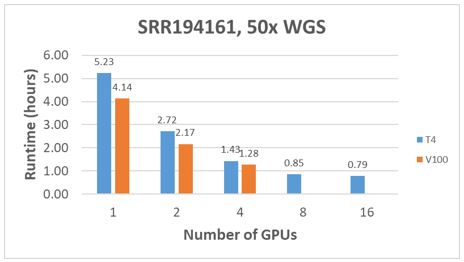
Figur 5 Sammenligninger af ydeevne med 50x WGS
Konklusion
En DSS 8440 med 16 T4 GPU'er er i stand til at behandle 30 50x menneskelige genomer pr. dag. En tilsvarende daglig analysekapacitet ved brug af en traditionel x86 CPU-arkitektur kræver ti PowerEdge C6420-databehandlingsnoder. Den komplette arkitektur diskuteres i en tidligere Dell-publikation.
Men hvis man dedikerer alle 16 T4 GPU'er til at behandle én prøve, er fordelen kun lille, da brugen af 16 GPU'er pr. analyse i bedste fald er 10 % hurtigere end brugen af 8 GPU'er. Designet af DSS 8440 tillader flere parallelle sekundære analyser. Ved at tildele otte T4 GPU'er pr. prøve øges den daglige analysekapacitet til ~50 genomer om dagen. Brug af fire GPU'er pr. prøve øger analysekapaciteten til ~70 genomer om dagen. Endnu vigtigere er, at denne daglige kapacitet ved brug af T4 GPU'er koster under det halve af brugen af et V100 GPU-design.
Ud over hastighed er kompatibilitet med andre analyseværktøjer afgørende for at kunne sammenligne resultaterne. Resultaterne af Parabricks-kimcelleanalysen er næsten identiske med den velkendte BWA-GATK Haplotype-bestemmelsesanalyse fra tidligere test. Vi ønskede også at sammenligne Parabricks-variantbestemmelsesresultaterne med andre værktøjssæt som f.eks. samtools/mpileup. Disse to helt forskellige værktøjer opnår ~90 % generel overensstemmelse for identificerede varianter, og variationer i mange velkendte genomikregioner, der indeholder vigtige gener, har en overensstemmelse på over 99 %.
Affected Products
DSS 8440, Isilon F800, Poweredge C4140, PowerEdge R640Article Properties
Article Number: 000180441
Article Type: How To
Last Modified: 22 Feb 2025
Version: 3
Find answers to your questions from other Dell users
Support Services
Check if your device is covered by Support Services.